発表のポイント
◆細胞内のタンパク質合成の途中で脱落したペプチジルtRNAを質量分析によって網羅的に解析する方法を構築しました。
◆タンパク質合成の初期段階では、誤った翻訳が頻繁に起きていること、また、それによって生じた誤ったペプチジルtRNAはリボソームから脱落する機構によって翻訳系から排除されることが明らかになりました。
◆これまでタンパク質合成の精度を維持する機構は、主にペプチド転移前に行われていると言われてきましたが、今回の研究によりペプチド転移後にも主要な校正機構があることが示されました。
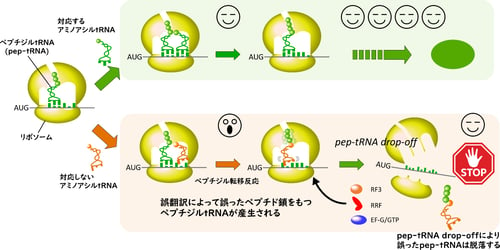
pep-tRNA drop-off機構によるペプチド転移反応後のタンパク質合成品質管理機構
発表概要
東京大学大学院工学系研究科の長尾翌手可講師と鈴木勉教授の研究グループは、大腸菌において、タンパク質合成の初期段階でリボソーム(注1)から脱落した伸長中のペプチジルtRNA(pep-tRNA)(注2)のペプチド鎖の配列を網羅的に解析することに成功しました。この解析により、タンパク質合成の初期段階では従来考えられていたよりもはるかに高い頻度で誤翻訳が起きていること、そして、その誤翻訳によって生じたpep-tRNAはタンパク質合成途中に積極的にリボソームから脱落し、翻訳の次のステップにもち越されないことを明らかにしました。その後、解離したリボソームは再び翻訳をやり直すことで、正しいタンパク質を作ることができます。この仕組みはタンパク質合成の品質を管理するための、これまでに知られていなかった全く新しい機構です。
バクテリアにおいて、タンパク質合成の初期段階では、翻訳伸長中のpep-tRNAが新生ペプチド鎖を結合したままリボソームから脱落するといったペプチジルtRNAドロップオフ(pep-tRNA drop-off)という現象が頻繁に起きています。この現象は1970年代には知られていましたが、技術的な障壁もあって、どのようなペプチド配列をもつtRNAが、どのようなmRNAから脱落しているのか、またその生物学的な意味については理解が進んでいませんでした。
本研究グループは、RNA単離技術と質量分析法を駆使し、大腸菌の全tRNA種について、脱落したpep-tRNAのペプチド鎖の配列同定を試みました。その結果、mRNAが指定する配列通りのペプチド鎖に加え、1アミノ酸だけ別のアミノ酸に置換されたペプチド鎖が大量に観察されました。その置換部位はペプチド鎖のC末端、つまりtRNAと結合しているアミノ酸残基に起きていることが判明しました。さらなる詳細な解析の結果、この誤った新生ペプチド鎖はリボソーム上で、tRNAがコドンを誤って解読し、ペプチド転移反応によって生じること、またその後、この誤ったpep-tRNAはリボソームから脱落し、翻訳の次のステップには進まないことが分かりました。
従来、翻訳精度を維持する機構としては、主に、リボソーム上でペプチド鎖が重合する前の段階、すなわちペプチド転移反応前でのみ起きていると考えられてきましたが、今回の発見は、ペプチド転移反応後にもタンパク質合成の品質を管理する主要な機構があることを意味し、従来の概念を完全に覆す発見と言えます。
発表内容
〈研究の背景〉
いついかなる時でも正確に遺伝情報通りのタンパク質を合成することは、生物に課せられた最も重要なタスクです。タンパク質合成は、アミノ酸を結合したtRNA(アミノアシルtRNA)が、リボソーム上でmRNAにコードされた遺伝暗号を正確に解読しながら進行します。その際の誤翻訳の頻度は5×103~6×104回に1回程度であり、一般に翻訳精度は高く保たれています。
バクテリアのタンパク質合成において、翻訳開始から約10アミノ酸残基程度までの伸長の初期段階では、約10%のpep-tRNAリボソームから脱落します。その後、リボソームは リボソームリサイクリング因子(RRF)と翻訳伸長因子であるEF-Gの作用によって解離し、再び翻訳をやり直します。脱落したpep-tRNAはペプチジルtRNAハイドロラーゼ(PTH)(注3)の作用により、ペプチド鎖とtRNA部分が切り離され、ペプチド鎖は分解され、遊離したtRNAは翻訳に再利用されます。PTHがバクテリアにとって必須遺伝子であり、その活性を失うと細胞内にpep-tRNAが急速に蓄積し、アミノアシルtRNAが枯渇することで死に至ります。したがって、pep-tRNA drop-offは生命の維持に関わる重要な機構であることが知られていました。しかし、その詳細な分子メカニズムや生理学的な役割については研究が進んでいませんでした。これは、細胞内のpep-tRNAを詳しく調べる解析手法がなかったことが大きな原因と考えられます。
〈研究の内容〉
細胞内のPTH活性は高く、野生型の大腸菌ではリボソームから脱落したpep-tRNAはすぐさま代謝されるため、その検出が困難でした。本研究グループは、まずPTHの温度感受性株(pthts)を作成し、非許容温度(42℃)で30分処理することで、PTHを失活させ、その細胞からpep-tRNAを低温かつ酸性条件下で抽出しました(図1A)。さらに本研究グループは、高速液体クロマトグラフィー質量分析法(LC/MS)(注4)を駆使することで、tRNAに結合しているアミノ酸や新生ペプチド鎖を網羅的に解析することに成功しました。実際に、5,500種類を超えるペプチド鎖(3-15アミノ酸残基)を検出しました。そして、各ペプチド鎖の精密質量を、大腸菌の全遺伝子のN末端領域のペプチド配列から作成した分子量リストと照らし合わせることで帰属を行いました。興味深いことに、pep-tRNA由来のペプチド鎖には、N末端ペプチドの1アミノ酸が置換されたペプチド鎖(non-cognate pep-tRNA)が大量に含まれていることがわかりました。実際に、検出したペプチド鎖の約20%がnon-cognate pep-tRNAであることが判明しています。
non-cognate pep-tRNAが産生される原因として、tRNAに誤ったアミノ酸が結合してしまうミスアミノアシル化と、tRNAが対応していないコドンを誤って解読してしまうミスコーディングが考えられました。これらの可能性について検証するために、pthts株から全種類のtRNAを往復循環クロマトグラフィー(注5)を用いて単離精製しました(図1A)。精製した各tRNAの3’末端に結合しているアミノ酸をLC/MSを用いて調べたところ、ミスアミノアシル化はどのtRNAにも検出されないことがわかりました。従って、non-cognate pep-tRNAはミスコーディングによって生じると考えました。
そこで、単離精製した各pep-tRNAから新生ペプチド鎖を調製し、LC/MS解析によりペプチド鎖の帰属を行ったところ、全種類のtRNAから合計で713個のペプチド鎖の配列を同定することに成功しました(図1B、C、D)。実際に、すべてのペプチド鎖は大腸菌遺伝子のN末端領域に帰属され、翻訳初期にpep-tRNAがリボソームから脱落するという機構を裏付ける結果となりました。このうちの約40%に相当する286個のペプチド鎖は1アミノ酸が置換したnon-cognate pep-tRNAであることが判明しました(図1D)。これらのペプチド鎖に関しては、アミノ酸の置換部位を調べたところ、ほぼすべてC末端、すなわちtRNAと結合している残基がmRNA上のコドンと対応しないアミノ酸であることが判明しました(図1B、C、D)。
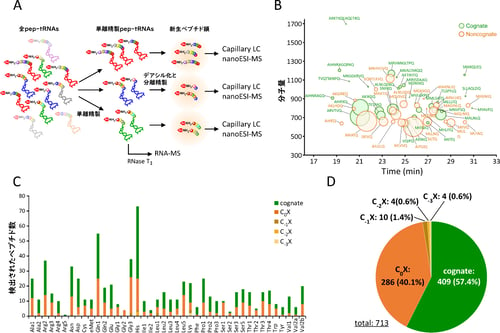
図1 質量分析によるアミノアシル、ペプチジルtRNAの解析
(A)pep-tRNAの調製および解析スキーム。 (B)tRNAGln1に結合していたペプチドの配列解析。
(C)全tRNA種における検出されたペプチド数。 CnXはnon-congnate pep-tRNAの種類。nはリボソームから脱落した部位からのアミノ酸置換の位置を示す。C0XはC末端で誤翻訳が起きているもの (D)配列同定を行ったpep-tRNA由来新生ペプチド鎖のタイプ別の集計。
本研究グループは、理化学研究所の岩崎信太郎主任研究員の研究グループと共同で、pep-tRNAがどのような分子機構でリボソームから脱落するかを解析したところ、RRFやペプチド解離因子RF3が関与することを明らかにしました(図2)。特に、RRFの機能を抑制した株(frrts)でリボソームプロファイリング(注6)を行ったところ、リボソームが各mRNAのN末端領域で顕著に停滞する様子が観察されました。この結果は、RRFが翻訳伸長の初期段階で積極的にpep-tRNAをリボソームから脱落させる機能を担っていることを示しています。
実際にミスコーディングがpep-tRNA drop-offを亢進させるか、について解析を行いました。ミスコーディングを誘発することが知られているtRNA修飾の欠損株(ΔtmcA)にレポーター遺伝子を発現させ、この遺伝子由来のpep-tRNAを解析したところ、ミスコーディングを誘発するとnon-cognate pep-tRNA の脱落が顕著に促進されることが判明しました。次に、ミスコーディングした部位よりも下流から脱落したpep-tRNAを調べたところ、アミノ酸が置換したペプチド鎖が顕著に減少していたことから、pep-tRNA drop-off機構はミスコーディングしたpep-tRNAをリボソームから脱落させることで翻訳の精度を保つ役割があることが判明しました。
では、なぜ翻訳初期段階にこのような誤翻訳が頻発するのでしょうか。近年、mRNAに使用されているコドンの使用頻度とその翻訳精度との関係が議論されています。一般的に、使用頻度が高いコドンを解読するtRNAは細胞内の存在量が多く、使用頻度の低いコドンを解読するtRNAは発現量が少ないことが知られています。従って、使用頻度が高いコドンは豊富に存在するtRNAによって速く正確に解読されるのに対し、使用頻度の低いコドンは少ないtRNAによって解読されるため、翻訳速度が遅く、また、他のtRNAによる競合によって翻訳精度が低下するリスクが高くなります。
そこで、大腸菌の各遺伝子(ORF)のN末端近傍(2~8アミノ酸残基)のコドン使用頻度を全体のコドン使用頻度と比較したところ、N末端近傍は、全体のコドン使用頻度とは異なる特徴をもっていることが判明しました(図2)。一方で、ORFの中央からC末端領域のコドンの使用頻度は、全体のコドン使用頻度とほぼ一致しています(図2)。以上の知見から、ORFのN末端近傍のコドン使用頻度は最適化されておらず、誤翻訳が頻繁に起きるのではないかと推察することができます。
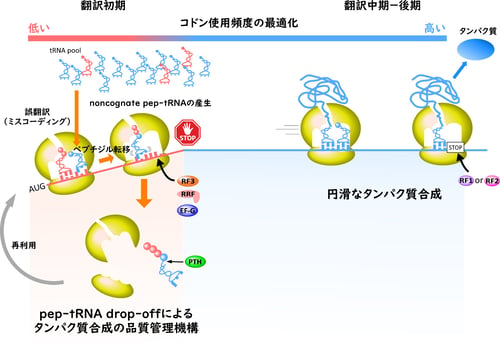
図2 pep-tRNA drop-offによる新規タンパク質合成の品質管理機構
バクテリアの翻訳初期段階では、その領域のコドン組成が翻訳システムに最適化されていないために誤翻訳が頻発し、翻訳精度が著しく低下する危険性があります。しかし、誤翻訳によって生じたnon-cognate pep-tRNAはdrop-off機構によって積極的に翻訳系から排除され、最終的な翻訳精度は高く保たれていると解釈できます(図2)。これまでの研究で、バクテリアの翻訳系は、主にイニシャルセレクション(注7)とプルーフリーディング(注8)と呼ばれる2つの機構によって、翻訳精度が高く維持されていることが知られていました。これらの機構はいずれもペプチド転移反応前の品質管理機構であり、ペプチド転移反応後には主要な品質管理機構は知られていませんでした。今回の研究成果により、ペプチド転移反応前の品質管理では排除しきれない誤翻訳を、ペプチド転移反応後にタンパク質合成の品質を管理する主要な機構があることが明らかとなり、従来の概念を完全に覆す発見と言えます。
〈今後の展望〉
今回の成果は、タンパク質合成の品質を管理する新しい仕組みを明らかにしたものであり、遺伝子発現の基礎研究に寄与するだけでなく、高品質な組換え体タンパク質の発現やバイオ医薬品の生産にもつながる成果と言えます。また、今後は哺乳動物や真核生物の翻訳系でも同様の品質管理機構が存在するかについて研究を進めていくことが重要です。
発表者
東京大学大学院工学系研究科化学生命工学専攻
長尾 翌手可(講師)
鈴木 勉(教授)
論文情報
〈雑誌〉Nature Communications
〈題名〉Quality control of protein synthesis in the early elongation stage
〈著者〉Asuteka Nagao, Yui Nakanishi, Yutaro Yamaguchi, Yoshifumi Mishina, Minami Karoji, Takafumi Toya, Tomoya Fujita, Shintaro Iwasaki, Kenjyo Miyauchi, Yuriko Sakaguchi, and Tsutomu Suzuki
〈DOI〉10.1038/s41467-023-38077-5
〈URL〉https://www.nature.com/articles/s41467-023-38077-5
研究助成
本研究は、日本学術振興会JSPSの基盤研究(S)「RNAエピジェネティックスと高次生命現象」(代表:鈴木勉、26220205)、基盤研究(S)「RNA修飾の変動と生命現象」(代表:鈴木勉、18H05272)、新学術領域研究 研究領域提案型「ncRNAのケミカルタクソノミ」(代表:鈴木勉、26113003)、新学術領域研究(研究領域提案型)「mRNAとタンパク質の品質管理機構における新生鎖の新規機能の解明」(代表:稲田利文、分担:長尾翌手可、14429723)、基盤研究(C)「tRNA使用頻度による新規翻訳調節機構の探索とメカニズムの解明」(代表:長尾翌手可、20266626)、および科学技術振興機構(JST)の戦略的創造研究推進事業(ERATO)「鈴木RNA修飾生命機能プロジェクト」(研究総括:鈴木勉、JPMJER2002)などの支援を受けて実施されました。
用語解説
(注1)リボソーム
RNAとタンパク質からなる複合体でタンパク質合成の場である。大小2つのサブユニットからなり、大サブユニットはペプチジル転移反応を、小サブユニットはmRNAとtRNA間のコドンーアンチコドン対合を監視するといった重要な役割をもつ。
(注2)ペプチジルtRNA(pep-tRNA)
Transfer RNA(転移RNA)はタンパク質合成の際に、mRNA上の遺伝暗号(コドン)に対応するアミノ酸を運ぶアダプター分子として働く。70~90塩基長の一本鎖RNAで、二次構造としてはクローバーリーフ様構造をとり、それが折りたたまれてL字型の立体構造をとる。アミノ酸が結合したtRNAをアミノアシルtRNA、ペプチド鎖が結合したtRNAをペプチジルtRNAと呼ぶ。
(注3)ペプチジルtRNAハイドロラーゼ(PTH)
リボソームから脱落したペプチジルtRNAのペプチド部分とtRNA部分を解離させる反応を触媒する酵素。PTHは多くのバクテリアで必須遺伝子である。
(注4)高速液体クロマトグラフィー質量分析法(LC/MS)
溶媒に溶けた物質を疎水性カラムなどによって分離し、質量分析装置によってその分離された物質をエレクトロスプレーイオン化法でイオン化し、その質量電荷比(m/z)を、経時的に計測する方法。ここではペプチドの質量を計測している。イオン化したペプチドをさらに質量分析計内で希ガスと衝突させることで分解し、生じたプロダクトイオンをもう一度スキャンすることで、ペプチドの配列決定が可能である。
(注5)往復循環クロマトグラフィー
本研究グループが独自に開発したRNA単離法。目的RNAと相補的な配列をもつDNAオリゴを結合させた樹脂にRNA試料を循環させることで、混在する試料から目的のRNAのみを単離精製することができる。
(注6) リボソームプロファイリング
細胞内のリボソームが翻訳しているmRNA領域を測定する方法。細胞から翻訳中のリボソーム画分を調製し、ヌクレアーゼ処理を行うと、リボソームがまさに翻訳していたmRNA領域(これをフットプリントと呼ぶ)のみが分解されずに残る。そのリボソームによって保護されたmRNA領域を次世代シークエンス法によって網羅的に測定することによって、細胞内で翻訳されているmRNA領域の特定やそのmRNAの翻訳量を知ることができる。
(注7) イニシャルセレクション
アミノアシルtRNAが伸長因子とGTPの三者複合体を形成し、リボソームのAサイトに運搬され、mRNAとの間のコドンーアンチコドン対合によってその対応関係をチェックする機構。もし対応が正しければ、アミノアシルtRNAは伸長因子から解離し、ペプチジル転移反応へと進むが、コドンーアンチコドン対合が間違っていれば、そのまま伸長因子とともにリボソームから解離する。タンパク質合成の精度を維持する上で非常に重要なチェック機構である。
(注8) プルーフリーディング
コドンと対応しないアミノアシルtRNAを排除するチェック機構の1つ。コドンーアンチコドン対合においてミスマッチが1か所だった場合、アミノアシルtRNAはイニシャルセレクションを通過してしまうことがあるが、ペプチジル転移反応に移行する過程でリボソームから解離し、翻訳系から排除される。イニシャルセレクションと並んで遺伝暗号解読の精度を左右する重要なチェック機構である。
プレスリリース本文:PDFファイル
Nature Communications:https://www.nature.com/articles/s41467-023-38077-5
おすすめ記事
本件に関連する記事はこちら
細菌の生存競争に関わるタンパク質の活性化の分子機構を解明 〜翻訳因子のこれまで知られていなかった新たな機能の発見〜
生物の耐熱性を支える「錠前」の発見 ~可逆的なリン酸化修飾がRNAを安定化する~


