発表のポイント
◆ヒト多能性幹細胞を用いて骨発生過程(軟骨内骨化)を再現する方法を確立しました。
◆再現したヒト骨組織に対して世界で初めてシングルセルマルチオーム解析を応用し、骨発生における転写制御ネットワークおよび転写因子ZEB2の機能について新たな知見を得ました。
◆本研究で得られた骨組織誘導法、シングルセルマルチオームデータおよび解析手法は、将来的に骨系統疾患の病態解明や新たな治療法の確立に役立つことが期待されます。
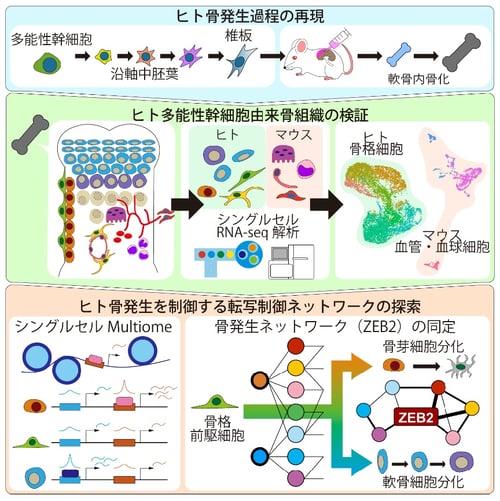
研究の概要図
発表概要
東京大学大学院医学系研究科の谷彰一郎日本学術振興会特別研究員、田中栄教授、北條宏徳准教授、東京大学大学院工学系研究科の鄭雄一教授、大阪大学大学院歯学研究科の大庭伸介教授(研究当時:東京大学大学院医学系研究科准教授、長崎大学生命医科学域(歯学系)教授)、米国コネチカット大学のDavid W. Rowe教授をはじめとする国際共同研究グループは、ヒト多能性幹細胞から作製した骨組織を用いて骨形成過程に寄与する重要な転写因子注1群を解明しました。
主にマウスを用いた研究手法によって、骨発生過程において重要な複数の転写因子が明らかにされてきましたが、細胞種ごとに異なる転写因子同士の関係性やヒト骨組織での検証は今まで十分ではありませんでした。本研究グループは、ヒト多能性幹細胞を用いた骨発生過程の再現と次世代シークエンサー(next-generation sequencer: NGS)を用いたシングルセル解析注2(scRNA-seq解析注3およびscATAC-seq注4を融合した最新のscMultiome解析注5)を駆使することで、ヒト骨発生過程における様々な転写因子の組み合わせ(転写制御ネットワーク注6)の一端を明らかにしました。本結果は、ヒト骨発生のメカニズムや骨系統疾患の病態理解への寄与と新たな治療標的の発見や治療戦略の確立へと発展することが期待されます。
発表内容
〈研究の背景〉
運動や内分泌、造血など様々な機能を有する骨組織は、3つの異なる由来(神経堤、側板中胚葉、沿軸中胚葉)から2つの異なる過程(膜内骨化、軟骨内骨化)を経て形成されます。我々の骨格の多くは中胚葉が起源であり、鋳型となる軟骨が一旦作られ、その後骨へと置き換わる(軟骨内骨化)ことで形成されます。この段階的な過程は、様々な細胞集団が特定の時期・場所ごとに機能することで成り立っていますが、その背景にあるメカニズムは完全には明らかになっていません。また、骨組織を構成する細胞(軟骨細胞や骨芽細胞)は共通の前駆細胞から分化することが知られているものの、細胞の運命が決定される仕組みは詳しくわかっていません。生体においてダイナミックに変化する細胞の状態(分化や機能)を理解するためには、その背景にある遺伝子発現とそれを制御する仕組みの理解が不可欠です。
同一個体内の細胞は、同一のゲノムDNA情報を共有していますが、そのDNA情報がどのように読み込まれ・書き出される(転写・翻訳)ことで機能を発揮するかは細胞毎に大きく異なり、これを制御する1つの機序が転写制御ネットワークになります。しかし、ヒトの骨発生過程における転写制御ネットワークについての理解は今まで不十分でした。加えて、骨組織に限らずヒトを対象とした研究領域では、倫理的な配慮が必要となります。そのため従来の研究では、主にマウスなどの実験動物を用いることで様々な知見が集積されてきました。しかしながら、実験動物で得られた知見をヒトへ応用する際の種差による再現性の問題も大きな課題です。一方で、近年iPS細胞に代表される多能性幹細胞は、無限の増殖能と多彩な分化能を背景に、様々な研究分野でヒト組織の構築や解析への応用が進んでいます。
〈研究の内容〉
研究グループは、ヒト多能性幹細胞を用いて骨発生過程を再現し、その背景にある転写制御ネットワークの解明に取り組みました。骨組織を作製するために、多能性幹細胞から沿軸中胚葉(椎板)を2次元培養で誘導した後、誘導した細胞をマウス体内へ移植する2段階の工程を用いました。得られた骨組織に対して組織学的解析とシングルセル解析を用いることで、骨組織を構成する様々な細胞集団を同定し、同時に重要な転写因子群の探索を行いました。
本研究ではまず、既存の椎板誘導法を簡略化(異種成分不含、組換タンパク製剤不含)し、現状で最も短期間(5日間)かつ簡便な分化誘導法を確立しました。誘導した細胞を免疫不全マウスの腎被膜下へ移植することで、約1-2か月で軟骨組織が形成され、3-4か月で軟骨内骨化と同様の骨組織像が確認されました。また、骨軟骨部分はヒト由来、血球はマウス由来であり、移植した椎板が骨軟骨を形成し、マウスの血管や血球を呼び込むことで骨髄構造(骨化中心)を形成したことが示唆されました。
次に、細胞移植後7週と19週の骨組織に対してscRNA-seq解析を行いました。その結果、軟骨内骨化過程における様々な細胞集団がヒト(骨軟骨)とマウス(血管・血球)の細胞で構成され、組織学的解析結果と矛盾しないことが示されました。さらに、ヒト胎児から取得された公共データとの統合解析を行い、本研究グループが誘導した骨組織との類似性が示されました。以上のことから、多能性幹細胞由来の骨組織が、ヒト胎児期の骨発生過程を部分的に再現していることが示唆されました。
そこで、オープンクロマチンおよび遺伝子発現解析を組み合わせたシングルセル多層解析(scMultiome)を誘導した骨組織に行いました。まず、遺伝子発現パターンから、scRNA-seq解析と同様の各骨格系細胞集団が同定され、データの整合性が示されました。続いて、各細胞集団におけるオープンクロマチン領域と遺伝子発現の関係を確認したところ、細胞種に特異的なオープンクロマチンと遺伝子発現の相関性が示されました。つまり、細胞種特異的なクロマチン構造の変化によって、細胞種特異的な遺伝子発現が制御されていることが示唆されました。さらに、オープンクロマチン領域における各転写因子のモチーフ解析注7を行い、遺伝子発現とモチーフの集積状況から、各細胞集団における各転写因子の活性を推定しました。その結果、細胞種毎に異なる転写因子群(転写制御ネットワーク)が活性化していることが示され、細胞の運命決定や機能発現において、これら複数の転写因子の組み合わせが重要であることが示唆されました。
最後に、上記の方法ではカバーしきれない転写因子についても、遺伝子発現の変動(RNA velocity解析注8)と発現の程度(DEG解析注9)を掛け合わせることで、細胞の状態変化により寄与していると推定される転写因子候補を探索しました。得られた候補の大半は既知の因子であり、この手法の妥当性が支持された一方で、骨形成において機能が明らかでない転写因子ZEB2を同定しました。ZEB2は骨芽細胞においてマスター転写因子であるRUNX2やSP7と共発現し、ZEB2をノックダウン(発現抑制)もしくはノックアウト(発現欠失)することで、骨分化に関連する遺伝子の発現が低下すること、そしてマウス胎生期の頭部を中心とした骨形成障害が明らかとなりました。以上の結果から、ZEB2が骨形成において複数の遺伝子発現を制御している(転写制御ネットワークの一端を担っている)ことが示されました。また、ノックアウトマウスにおける表現型は、ZEB2の機能障害が原因として知られているMowat-Wilson症候群の頭部骨格異常と類似しており、骨芽細胞でのZEB2の機能不全がその一因である可能性も示唆されました。将来的な骨系統疾患における病態解明や新たな治療標的の発見についての1つの可能性を示したものと考えられます。
〈今後の展望〉
本研究では、ヒトの軟骨内骨化の再現、その過程における各骨格細胞の遺伝子発現およびオープンクロマチン領域情報の取得、運命決定や分化過程を制御しうる転写制御ネットワークの同定が行われました。一連のNGSデータは公共データベースへ登録されております。また、本研究が世界で初めて、ヒト多能性幹細胞から作製した骨組織に対してscMultiome解析を応用しました。以上の結果は、ヒトの骨発生過程を研究するための貴重なリソースを提供するだけなく、細胞の状態に応じてダイナミックに変化する転写制御ネットワークの理解、そして正常な骨発生過程および骨系統疾患の機序解明や新規治療標的の発見に役立つ成果であると考えられます。
発表者
東京大学
大学院医学系研究科 外科学専攻 感覚・運動機能医学講座
谷 彰一郎(日本学術振興会特別研究員)
田中 栄(教授)
大学院医学系研究科 附属疾患生命工学センター 臨床医工学部門
北條 宏徳(准教授)〈東京大学大学院工学系研究科 バイオエンジニアリング専攻〉
大学院工学系研究科 バイオエンジニアリング専攻
鄭 雄一(教授)
〈東京大学大学院医学系研究科 附属疾患生命工学センター 臨床医工学部門〉
大阪大学大学院歯学研究科
大庭 伸介(教授)
〈研究当時:東京大学大学院医学系研究科附属疾患生命工学センター(准教授)、長崎大学生命医科学域・歯学系(教授)〉
論文情報
〈雑誌〉Cell Reports(オンライン版:3月24日11時 ※米国東部夏時間)
〈題名〉Stem-Cell-Based Modeling and Single-Cell Multiomics Reveal Gene Regulatory Mechanisms Underlying Human Skeletal Development
〈著者〉Shoichiro Tani*, Hiroyuki Okada, Shoko Onodera, Ryota Chijimatsu, Masahide Seki, Yutaka Suzuki, Xiaonan Xin, David W. Rowe, Taku Saito, Sakae Tanaka, Ung-il Chung, Shinsuke Ohba*, and Hironori Hojo*(*責任著者)
〈DOI〉10.1016/j.celrep.2023.112276
〈URL〉https://www.cell.com/cell-reports/fulltext/S2211-1247(23)00287-5
研究助成
本研究は、主に科研費「基盤(A)(課題番号:21H04952)」、「基盤(B)(課題番号:20H03885)」、「基盤(B)(課題番号:17H04403)」、AMED「再生医療実現拠点ネットワークプログラム(課題番号:JP21bm0704071)」の支援により実施されました。
用語解説
(注1)転写因子RUNX2、SP7、ZEB2
ゲノムDNAのプロモーター領域やエンハンサー領域に結合して、標的とする遺伝子の転写量の調節にはたらくタンパク質を転写因子と呼ぶ。遺伝子改変マウスを用いた解析により、Runx2やSp7遺伝子がないと骨発生が起こらないことから、骨のマスター転写因子と呼ばれている。ZEB2はzinc finger E-box binding homeobox 2の略で、上皮間葉転換において重要な働きをする因子で、がんや神経の領域でよく研究されている。ZEB2遺伝子が半分機能を失うことで、精神や神経、骨格を含めた様々な症状をきたすMowat-Wilson症候群という疾患を引き起こすことが知られている。
(注2)シングルセル解析(Single-cell analysis)
従来はまとまった細胞集団に対しての解析が主流であったが、異なる細胞集団の平均値としての結果しか見ることができず、そこに含まれる様々な細胞について十分な解像度のデータ取得が困難であった。近年、各1細胞におけるデータ取得が技術革新によって可能になった。
(注3)シングルセルRNAシークエンス解析(scRNA-seq:single-cell RNA sequencing)
1細胞毎にRNA情報から遺伝子発現データを取得できる解析手法。
(注4)シングルセルATACシークエンス解析(scATAC-seq:single-cell RNA sequencing)
ATACはAssay for Transposase-Accessible Chromatin-シークエンシング法の略語。Tn5 トランスポゼースを用いてオープンクロマチン領域の断片化し、ゲノム全域でオープンクロマチン構造を選択的に検出することが可能になる。
(注5)シングルセル多層解析(scMultiome)
今回はscRNA-seqとscATAC-seqを統合して実施。1細胞毎に遺伝子発現およびオープンクロマチン領域情報が取得可能になる。
(注6)転写制御ネットワーク(Gene regulatory networks(GRNs))
各転写因子は、下流の転写因子の発現を制御しているが、その下流の転写因子もさらにその下流の因子を制御するというようにして互いに制御しあう関係があり、このネットワークのような関係性によって特定の機能や挙動を細胞にもたらすと考えられている。今回は転写因子の発現とモチーフ活性からこのネットワークの中核となる因子をscMultiome解析によって推定した。また、ZEB2の機能解析によって、これら複数の転写因子と制御関係にあることが示されたため、ZEB2自体も骨におけるこのネットワークの一員であると示唆された。
(注7)モチーフ解析
各転写因子は特定の配列(モチーフ)を持ったDNA領域に結合し、この配列は転写因子ごとに異なることが知られている。オープンクロマチン領域において、このモチーフがどの程度みられるかによってどの転写因子がクロマチン構造の変化(とそれに伴う遺伝子発現の変動)に寄与しているかを推定することが可能である。
(注8)RNA velocity解析
遺伝子情報が読み取られてタンパク質へと変換される工程において、mRNA(メッセンジャーRNA)がその鋳型となっている。mRNAはDNAから複製される段階で当初不要な部分(イントロン)を含んでいるため、これが途中でスプライシングされ、タンパク質に必要な部分(エキソン)のみになる。そのため、このスプライシング前後のmRNAの比率を調べることで、その遺伝子の発現が増えている途中か減っている途中かを推定することが可能になる。この増減は細胞の状態変化(分化を含む)を反映していると考えられるため、ある時点での遺伝子発現単独の情報よりも細胞の動態を反映しているものと考えられる。
(注9)DEG解析(Differentially expressed gene analysis)
対象となる細胞集団において、他のグループと比較してより発現が高い・低い遺伝子を算出する手法。これによって、異なる細胞集団において特徴的な遺伝子を効率的に検出することが可能になる。
プレスリリース本文:PDFファイル
Cell Reports:https://www.cell.com/cell-reports/fulltext/S2211-1247(23)00287-5
おすすめ記事
本件に関連する記事はこちら
ヒストンH3タンパク質のクロトニル化修飾により 大腸がん細胞が増幅する仕組みを解明 〜⽴体構造解析に基づく大腸がんの新治療薬開発に期待〜
骨形成における新しい遺伝子制御メカニズムとその進化学的意義 ~骨形成に必須の転写制御因子Sp7/osterixの作動様式が明らかに~ : バイオエンジニアリング専攻 大庭伸介特任准教授ら