1.発表者
上野 博史 (東京大学 大学院工学系研究科 応用化学専攻 助教)
野地 博行*(東京大学 大学院工学系研究科 応用化学専攻 教授)
*:責任著者
2.発表のポイント
◆クライオ電子顕微鏡(注1)を用いた単粒子解析により、これまで構造が未解明であったATP合成酵素のF1モーターの基質(ATP)結合待ちの構造を明らかにしました。
◆これまでの1分子回転解析(注2)で予測されていたF1モーターの6つの回転中間体の構造全てが明らかになり、6ステップで動くF1モーターの回転の分子描像が完全に明らかになりました(図2)。
◆今回の成果は、既存の分子モーターが関わる生命機能の理解だけでなく、新たな分子モーターのデザイン・創成につながると期待されます。
3.発表概要
我々も含めすべての生物は、アデノシン3リン酸(ATP)の加水分解で得られるエネルギーを用いて生命活動を維持しています。そのためATPは細胞のエネルギー通貨と呼ばれています。生命を維持するためのATPの大部分を産生しているのは、ATP合成酵素と呼ばれるタンパク質複合体です。ATP合成酵素は、FoモーターとF1モーターと呼ばれる2つの回転分子モーターが結合した複合体で(図1)、ATP合成反応はF1モーターが担います。
F1モーターは、単体ではATP合成の逆反応を触媒します。すなわち、ATPを加水分解しながらその化学エネルギーを用いて、F1モーター分子内部の回転子をATP合成時とは逆方向に回転させます。F1モーターは、化学エネルギーと回転運動を相互変換するエネルギー変換素子とみなすことができ、そのエネルギー変換効率が100%に近いことから生物学にとどまらず物理・化学の分野からもそのメカニズムが注目されています。
今回の発表はF1モーターの新しい構造状態に関するものです。F1モーターは、3種類のタンパク質(α, β, γ)が3:3:1の割合で自己集合し、α3β3固定子リングに回転子γが突き刺さった構造を持ちます。ATP加水分解/合成の触媒部位はβサブユニットに存在し、ATP加水分解時には、このβサブユニットがATPの加水分解に伴い構造変化をすることでトルクを発生し回転子であるγを回転させています。
これまで、私たちはF1モーターの回転の様子を1分子解析することで、そのメカニズムを詳細に解析してきました。その結果、βサブユニットが6つの状態を経ることでF1モーターが1回転することが分かっていました(図2左)。一方で、これまでのF1モーターの構造解析ではそのうちの3つの状態しか確認されておらず、残りの3つの状態がどのような構造なのか明らかではありませんでした。
今回、東京大学大学院工学系研究科の上野博史助教、野地博行教授らは、オーストラリアのVictor Chang Cardiac Research Instituteの共同研究者らと共に、クライオ電子顕微鏡を用いた単粒子解析により、βの残りの3つの状態を示したF1モーターの構造を解析することに世界で初めて成功しました。これによって、βがトルクを発生するときの構造変化の様子を6つのスナップショットとして示すことに成功しました。加えて、ATP加水分解で生じる反応生成物(リン酸)がタンパク質から出ていく出口構造も初めて発見することができました(図3)。これらの結果は、回転分子モーターの分子描像を完全に明らかにしただけではなく、新たな分子モーターの人工設計や創成につながると期待されます。
4.発表内容
・研究の背景
ATP合成酵素は、我々の細胞内のミトコンドリアや微生物の原形質膜などに存在し、細胞のエネルギー通貨であるアデノシン3リン酸(ATP)の大部分を合成しています。このATP合成酵素は、膜に埋まったFoモーターと、膜から突き出たF1モーターの2つの回転分子モーターが結合した複合体です(図1)。Foモーターは膜内外に形成されたイオンの電気化学的エネルギーを用いて、F1を回転させATPを合成します。一方、F1モーター単体ではATP加水分解の化学的エネルギーを利用して、ATP合成時とは逆方向に回転します。つまり、F1モーターは、化学エネルギーと回転運動を相互変換するエネルギー変換素子とみなすことができます。これまで私たちは主に好熱菌Bacillus PS3由来のF1(TF1)を用いた1分子回転解析を行うことで、そのメカニズムを詳細に解析してきました。その結果、このF1は3つの基質(ATP)結合待ち構造と3つの基質分解待ち構造、合計6つの状態を経ることで1回転することが分かっていました(図2左)。一方、F1の回転に伴う化学力学共役機構の詳細な理解のため回転と構造との対応づけを行おうと、原子レベルで立体構造を明らかにできるX線結晶構造や電子顕微鏡解析も盛んに行われてきました。しかしながら、これまでは基質分解待ちの構造しか得られておらず、1分子回転解析で観察されている基質結合待ちの状態の構造は明らかではありませんでした。
・研究の内容
上述の背景のもと、私たちは共同研究者のAlastair Stewart博士らのグループと共に、クライオ電子顕微鏡を用いた単粒子解析によるTF1の基質結合待ち構造の解明を目指しました。基質結合待ち構造の取得には、ATPを加えた後、反応中のF1を基質結合待ちの構造に長時間留めておくことが求められます。そこで私たちは低温で基質結合待ち状態に陥る変異F1を用いて試料を作製することで、その実現を試みました。その結果、これまで得られていない基質結合待ちの構造を得ることに成功しました。またこの変異F1は、基質結合以外にも基質分解が遅くなっているため、基質分解待ち構造も得られることが期待されました。実際、得られた単粒子をクラス分けすることにより基質結合に加えて基質分解待ちの構造を得ることにも成功しました。つまり、これまでの1分子回転計測で予測されていたTF1モーターの6つの回転中間体の構造全てが本研究で明らかになったと言えます(図2右)。これらの構造から、これまで報告されていない触媒サブユニットβの新規構造や、ATP加水分解で生じる反応生成物(リン酸)解離のパスを発見することにも成功しました(図3)。これらの結果から、βがトルクを発生するときの構造変化の様子を6つのスナップショットとして示すことにも成功しました。
・今後の展望
回転分子モーターの回転中の分子描像を完全に明らかにした今回の研究は、既存の分子モーターの化学・力学共役の理解だけでなく、そこから得られた知見をもととした新たな分子モーターのデザイン・創成につながると期待されます。またクライオ電子顕微鏡を用いて、分子モーターの反応中の状態をクラス分けで複数とらえるという今回の研究手法は、分子モーター研究の推進に非常に有用であり、様々な既存の分子モーター、新規分子モーターに応用されていくと期待されます。
・本研究の助成事業
本研究は、日本学術振興会(JSPS)科学研究費助成事業 新学術領域研究(研究領域提案型)(課題番号:18H04817, 19H05380、研究代表者:上野博史) の支援を受けて行われました。
5.発表雑誌
雑誌名: Nature Communications (オンライン版:2021 年 8 月 3 日)
論文タイトル: The six steps of the complete F1-ATPase rotary catalytic cycle
著者: Meghna Sobti, Hiroshi Ueno, Hiroyuki Noji*, Alastair G. Stewart*
(*:責任著者)
DOI 番号: 10.1038/s41467-021-25029-0
6.用語解説
注1:クライオ電子顕微鏡
透過型電子顕微鏡の一種。試料を極低温の薄い氷に閉じ込めて液体窒素(-196℃)冷却下で観察を行う。そのためタンパク質などの生体分子に対しての電子線のダメージが軽減される。タンパク質の立体構造を高分解能で決定する手法として近年目覚ましい進歩を遂げている。
注2:1分子回転解析
分子モーターの回転を光学プローブを用いて直接光学顕微鏡下で観察し解析する手法。F1モーターでは、1997年に回転の直接観察に初めて成功した後(Noji et al., Nature 1997)、高時間分解能での解析、変異体の解析および基質結合・解離と回転の同時観察などによりその回転機構の詳細が明らかになっている。
7.添付資料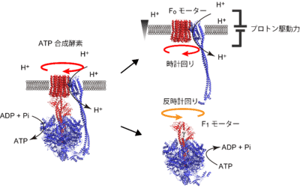 (図1)ATP合成酵素
(図1)ATP合成酵素
ATP合成酵素はプロトン駆動力で駆動されるFoモーターとATP加水分解で駆動するF1モーターからなる。F1モーターは固定子であるα3β3リング(青色)の中央に回転子γサブユニット(赤色)が突き刺さった構造を取る。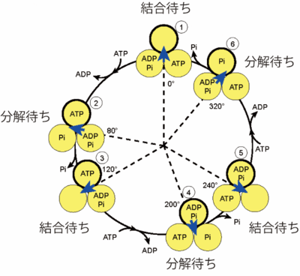
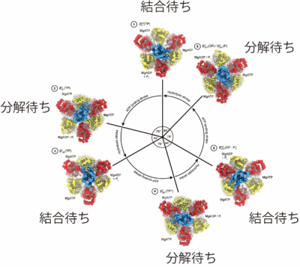
(図2)1分子回転解析から提唱された回転スキーム(左)と明らかになった構造(右)
触媒サブユニットβを丸で、回転するγサブユニットの向きを矢印で示す。各βサブユニットの化学状態を示している(左)。回転スキームと明らかになった構造とを対応づけたものを右に示す。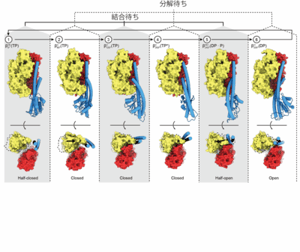
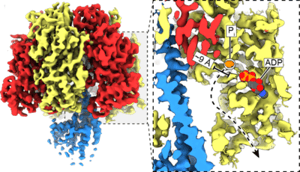 (図3)各角度での触媒サブユニットβの構造(上)とリン酸解離トンネル(下)
(図3)各角度での触媒サブユニットβの構造(上)とリン酸解離トンネル(下)
図2の①~⑥までのβサブユニットの構造を上に示す。下は新規に発見したリン酸解離トンネル。
プレスリリース本文:PDFファイル
Nature Communications:https://www.nature.com/articles/s41467-021-25029-0
おすすめ記事
本件に関連する記事はこちら

