兵庫県立大学大学院理学研究科の澤井仁美助教を中心とした共同研究グループは、大型放射光施設「SPring-8」[1]を利用して、病原菌がヒトなどの動物の血液から栄養として獲得した「ヘム」[2]の濃度を制御するために用いる「ヘム濃度センサータンパク質」の立体構造を決定し、世界で初めてその作動機序を原子レベルで解明しました。この結果により、病原菌が感染先(宿主)の体内で生き残っていくために利用するシステムへの理解が進み、このシステムをターゲットにした新たな抗菌剤開発に貢献できる可能性があります。本研究の成果は、2021年4月13日の日本時間18時付で、Nature Publishing Groupが発行する国際生物科学雑誌『Communications Biology』に掲載されました。
1.研究の背景
鉄は、全ての生物にとって必須の金属元素であり、生物体内で種々の重要な反応に関わっています。例えば、ヒトでは呼吸で肺に取り込まれた酸素を末梢組織に運ぶ赤血球の中のヘモグロビン[3]に、鉄がヘムの形で多量に含まれています。病原菌が生きて増殖するためにも鉄が必須です。ペスト菌・コレラ菌・ジフテリア菌・炭疽菌などの病原菌は、他の生物からヘムを奪ってその中の鉄を利用するシステムを進化させています。そのような病原菌の一種である溶血性連鎖球菌[4]は、ヒトなどの動物の血液に入り込み、赤血球を破壊してヘモグロビンを放出させ、ヘモグロビンに含まれるヘムを菌体内に取り込んで栄養として利用するシステムを有しています(図1)。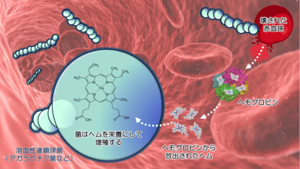 図1: 溶血性連鎖球菌が宿主動物の赤血球から栄養としてヘムを獲得するシステム
図1: 溶血性連鎖球菌が宿主動物の赤血球から栄養としてヘムを獲得するシステム
1個の赤血球を壊すと10億分子以上の大量のヘムが放出されるので、この病原菌はそのヘムを栄養として利用して生きています。しかし、菌体内で余剰になったヘムは、活性酸素[5]を発生させるため、病原菌にとっては「毒」となります。この余剰なヘムによる毒性を回避するために、病原菌は菌体内のヘムの濃度を感知して菌体外へ排出するシステムを有していることが、近年報告されています。この「ヘム毒性回避システム」は病原菌が宿主内で生存するために必須のシステムと考えられていますが、病原菌が余剰なヘムによる毒性をどのようにして回避しているのかは未解明でした。
2.研究の手法と成果
本研究では、新生児の敗血症・髄膜炎・肺炎や妊婦の子宮内膜炎の原因となる溶血性連鎖球菌(アガラクチア菌Streptococcus agalactiae)がもつPefRというタンパク質を研究対象にしました。PefRは、通常は遺伝子(DNA)に結合しているタンパク質です。しかし、菌体内に余剰なヘムが増えてくると、PefRはそのヘムと結合してDNAから外れます。PefRがDNAから外れることをスイッチにして、余剰なヘムを菌体外に排出するタンパク質「ヘム排出タンパク質」が作られます(図2)。このように、PefRは菌体内のヘム濃度を感知するための「ヘム濃度センサータンパク質」として機能する転写調節因子[6]として働いています。つまり、PefRは病原菌がもつ「ヘム毒性回避システム」のキープレイヤーであるといえます。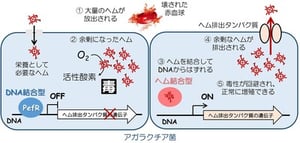 図2: アガラクチア菌のヘム濃度センサータンパク質PefRの機能
図2: アガラクチア菌のヘム濃度センサータンパク質PefRの機能
左の青い四角は栄養として必要なヘムしか存在しない時のアガラクチア菌をあらわしている。この時、PefRはヘム排出タンパク質の遺伝子の上流に結合してヘム排出タンパク質を作らせないようにしている。アガラクチア菌に赤血球が壊されて大量のヘムが放出される(①)と、余剰なヘムが菌体内に流入する(②)。右の青い四角は余剰なヘムが増えた時のアガラクチア菌を示している。この時、PefRが余剰なヘムを結合しDNAからはずれ(③)、ヘム排出タンパク質の合成が促進する。ヘム排出タンパク質によって余剰なヘムは菌体外へと排出され(④)、毒性が回避されてアガラクチア菌は正常に増殖できるようになる(⑤)。
本研究グループはPefRがヘムを感知してDNAからはずれる仕組みを明らかにするために「DNA結合型」と「ヘム結合型」などの各状態で単結晶を調製し、大型放射光施設「SPring-8」のタンパク質結晶構造解析用ビームライン(BL26B2, BL41XU, BL44XU)の高輝度なX線ビームを利用して、世界で初めてPefRの立体構造を解明しました。その結果、菌体内のヘムが少なく、ヘムを結合していないPefRはDNAを挟み込んでまたがるような形(構造)で存在していることがわかりました。ヘム濃度が上がり、ヘムがPefRに入って結合すると、それを引き金にしてPefRのDNAを挟み込む領域の間の距離が広がってしまい、もはやDNAを挟み込めない構造に変わっていました。PefRがはずれたDNAにはヘム排出タンパク質を作る設計図(遺伝情報)が書かれていますので、それに従ってヘム排出タンパク質が作られ、余剰のヘムを菌体外に排出するという病原菌の「ヘム毒性回避システム」の作動機序を解明しました(図3)。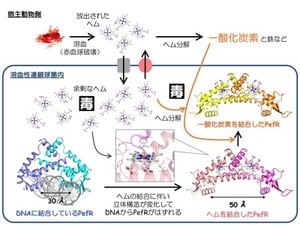 図3: 本研究で明らかにした3つの状態でのPefRの立体構造とそれらの存在意義
図3: 本研究で明らかにした3つの状態でのPefRの立体構造とそれらの存在意義
DNA結合型PefRの立体構造におけるタンパク質領域は青色と水色、DNA領域は灰色で描いている。ヘム結合型PefRはピンク色とマゼンタ色、一酸化炭素結合型PefRは黄色とオレンジ色で示している。PefRが菌体内で余剰になって遊離しているヘムを結合すると、DNAに結合できない構造に変化する。さらにヘム結合型PefRは、宿主動物または菌体内でのヘム分解反応により生じた一酸化炭素を結合する性質を備えている。
さらなる生化学的な解析により、ヘムを結合して一旦DNAから離れたPefRは、一酸化炭素を安定に結合でき、この一酸化炭素結合型PefRはもはやDNAとは再び結合できず菌体内で一酸化炭素を捕捉できることを発見しました(図3)。一酸化炭素は、宿主動物の体内でヘム分解反応の生成物として放出されますが、それが病原菌体内に流入すると微量でも「毒」となり菌体の死滅や休眠を招いてしまいます。PefRは余剰になったヘムの毒性だけでなく、一酸化炭素による毒性も回避する働きを果たしていることを示しています。
3.この研究の社会的意義と今後の展望
近年、薬剤耐性菌が蔓延し、抗菌薬が効かなくなることが問題になっています。世界保健機関WHOは、抗菌薬が効かなくなって2050年には年間1千万人が細菌感染によって死亡すると予測しています。既存の抗菌薬の開発は、病原菌の細胞膜・細胞壁・核酸・タンパク質の合成の阻害をターゲットとしており、新規抗菌薬の開発は、現在のところ停滞しています。病原菌のヘム獲得システムは、病原菌の生育に必須であるため、新たな抗菌薬のターゲットになりえます。本研究で明らかにした「ヘム濃度センサータンパク質」の立体構造や性質は、新たな抗菌薬の開発に貢献できる可能性があります。
4.用語説明
[1] SPring-8: 大型放射光施設「SPring-8」は兵庫県の播磨科学公園都市にある世界最高性能の放射光を生み出す理化学研究所の施設。SPring-8の名前はSuper Photon ring-8 GeV(80億電子ボルト)に由来。放射光とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げた時に発生する強力な電磁波のこと。放射光を用いてナノテクノロジー・バイオテクノロジー・産業利用まで幅広い研究が行われている。
[2] ヘム: ポルフィリンと呼ばれる環状平面分子の中心に鉄原子をもつ化合物。ポルフィリン環の修飾の種類や位置によっていくつかの種類に分類される。ヘムを分子中に取り込んで、はじめてその機能が発揮されるタンパク質をヘムタンパク質と呼び、通常、赤色を呈する。酸素運搬体であるヘモグロビン、電子伝達に関与するシトクロム類、酵素活性をもつペルオキシダーゼなどがヘムタンパク質の代表例。本研究対象のPefRは、細胞内に存在するヘムを感知する「ヘム濃度センサータンパク質」に分類される。
[3] ヘモグロビン: 脊椎動物の血液中の赤血球にたくさん含まれるヘムタンパク質。1分子あたりヘムを4分子結合しており、それらのヘムに酸素分子を結合して全身に酸素を運搬している。赤血球中のヘモグロビンに結合しているヘムが赤いので、脊椎動物の血液は赤くみえる。
[4] 溶血性連鎖球菌: 溶血とは血液中の赤血球が壊される現象。溶血性連鎖球菌は、球状の細胞が連なった形態をとる連鎖球菌の中でも溶血作用を示すもの。ヘムの生合成遺伝子を欠損しており自らでヘムを生合成できないため、宿主動物の血液に感染して溶血を引き起こし、赤血球を壊してヘムを獲得する。本研究対象のPefRを有するアガラクチア菌 (Streptococcus agalactiae)の他にも、肺炎球菌 (Streptococcus pneumoniae)、化膿レンサ球菌(溶連菌、Streptococcus pyogenes)、ミュータンス菌(Streptococcus mutans)などが知られている。
[5] 活性酸素: 大気中に含まれる酸素分子が化学的に反応性の高い状態に変化した化学種の総称。・OH(ヒドロキシラジカル)、O2・-(スーパーオキサイドアニオンラジカル)、H2O2 (過酸化水素)などがある。DNA、脂質、タンパク質などの生体分子に酸化ダメージを与え、本来の生理機能を失わせる作用がある。
[6] 転写調節因子: 転写制御因子あるいは転写因子ともいう。DNAに配列特異的に結合するタンパク質の一群。DNAに結合し、RNAを合成する酵素によるDNAの転写を活性化あるいは不活性化する。本研究対象のPefRは、DNAに結合することで転写を不活性化するタイプの転写調節因子である。
5.論文情報
掲載誌: Communications Biology (Nature Publishing Groupが発行する国際生物科学雑誌)
論文タイトル: Heme Controls the Structural Rearrangement of Its Sensor Protein Mediating Bacterial Survival
(ヘムが溶血性細菌の生存に関係するセンサータンパク質の構造変化を制御する)
著者: Megumi Nishinaga, Hiroshi Sugimoto, Yudai Nishitani, Seina Nagai, Satoru Nagatoishi, Norifumi Muraki, Takehiko Tosha, Kouhei Tsumoto, Shigetoshi Aono, Yoshitsugu Shiro, Hitomi Sawai
DOI: 10.1038/s42003-021-01987-5
掲載日時: 2021年4月13日 日本時間18時(ロンドン時間10時)
6.研究グループ
兵庫県立大学大学院 理学研究科
教授 城 宜嗣(しろ よしつぐ)
助教 澤井 仁美(さわい ひとみ)
非常勤研究員 西谷 雄大(にしたに ゆうだい)
大学院生 [研究当時] 西永 惠(にしなが めぐみ)
学部生 [研究当時] 長井 聖奈(ながい せいな)
理化学研究所 放射光科学研究センター 利用システム開発研究部門 生物系ビームライン基盤グループ 生命系放射光利用システム開発チーム
専任研究員 杉本 宏(すぎもと ひろし)
専任研究員 當舍 武彦(とうしゃ たけひこ)
東京大学 大学院工学系研究科 バイオエンジニアリング専攻ならびに化学生命工学専攻
教授 津本 浩平(つもと こうへい)
東京大学医科学研究所 先進的バイオ医薬品学社会連携研究部門
特任准教授 長門石 曉(ながといし さとる)
自然科学研究機構 分子科学研究所
教授 青野 重利(あおの しげとし)
助教 村木 則文(むらき のりふみ)
7.研究サポート
本研究は、文部科学省科学研究費補助金の基盤研究 (S) (課題番号26220807)、基盤研究(B) (課題番号18H02396)、基盤研究 (C) (課題番号18K05321)、新学術領域研究「生命金属科学」(課題番号19H05761)の一環として行われました。さらに、公益信託山村富美記念女性自然科学者研究助成基金、公益財団法人ひょうご科学技術協会、理化学研究所の新領域開拓課題「分子システム研究」ならびに独創的研究課題「脂質の統合的理解」と「物質階層の原理を探求する統合的実験研究」、国立研究開発法人日本医療研究開発機構 (AMED) 創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム (BINDS)(課題番号 JP20am0101094)による支援を受けて進められました。
プレスリリース本文:PDFファイル
Communications Biology : https://www.nature.com/articles/s42003-021-01987-5
理化学研究所:https://www.riken.jp/press/2021/20210414_1/index.html
分子科学研究所:https://www.ims.ac.jp/news/2021/04/14_4939.html
日本経済新聞:https://www.nikkei.com/article/DGXLRSP608500_T10C21A4000000/
おすすめ記事
本件に関連する記事はこちら
伝導電子と局在スピン・軌道が織りなす悪魔の調律~多極子の衣をまとった電子「多極子ポーラロン」を発見~
悪魔と取引した電子たち~磁性体における40年来の謎を解明~:物理工学専攻 有田 亮太郎教授ら